Bacteria classification bằng thư viện fastai
Bài đăng này đã không được cập nhật trong 5 năm
Giới thiệu
fastai là 1 thư viện deep learning hiện đại, cung cấp API bậc cao để giúp các lập trình viên AI cài đặt các mô hình deep learning cho các bài toán như classification, segmentation... và nhanh chóng đạt được kết quả tốt chỉ bằng vài dòng code. Bên cạnh đó, nhờ được phát triển trên nền tảng thư viện Pytorch, nên fastai còn cung cấp các cấu phần bậc thấp cho các nhà nghiên cứu phát triển mô hình mới, cũng như hoàn toàn tương thích với các thành phần của pytorch.
Trong bài viết này, mình sẽ giới thiệu về 1 số tính năng của fastai và áp dụng chúng để xây dựng 1 mô hình phân lớp. Let's get started !!!
Cài đặt fastai
Các bạn có thể cài đặt fastai trên máy mình bằng câu lệnh sau:
pip install fastai --upgrade -q
Sau khi cài đặt thì chạy đoạn code sau để import fastai và các thư viện cần thiết:
import os
import requests
import urllib.request
import zipfile
import matplotlib.pyplot as plt
from torchsummary import summary
from fastai.vision.all import *
from bs4 import BeautifulSoup
Khi import fastai thì một số thư viện phổ biến như numpy, pandas, matplotlib cũng được import cùng nên không cần import lại nữa
Dữ liệu
Mình sẽ sử dụng bộ dữ liệu ảnh chụp vi khuẩn lấy từ trang web này. Các bạn có thể tải dữ liệu thẳng từ trên website về máy sau đó giải nén hoặc nếu ai dùng google colab thì có thể dùng đoạn code này:
os.makedirs('dibas_zip')
os.makedirs('dibas_images')
url = 'http://misztal.edu.pl/software/databases/dibas/'
response = requests.get(url)
soup = BeautifulSoup(response.text, "html.parser")
links = [tag['href'] for tag in soup.findAll('a')]
for link in links:
if ".zip" in link:
file_name = link.partition("/dibas/")[2]
urllib.request.urlretrieve(link, 'dibas_zip/' + file_name)
zip_ref = zipfile.ZipFile('dibas_zip/' + file_name, 'r')
zip_ref.extractall('dibas_images/')
zip_ref.close()
print("Downloaded and extracted: " + file_name)
Dữ liệu của chúng ta gồm 692 ảnh:
fns = []
for root,dirs,files in os.walk(path, topdown=true):
for f in files:
fns.append(root/Path(f))
len(fns), fns[0]
![]()
Tạo Dataloader
fastai cung cấp API cho việc tạo Dataloader của pytorch 1 cách đơn giản và nhanh chóng
dblock = DataBlock(blocks=(ImageBlock, CategoryBlock),
get_y=RegexLabeller(r'/(.+)_\d+.tif$'),
splitter=RandomSplitter(valid_pct=0.1),
item_tfms=[Resize(512)])
Đoạn lệnh trên sẽ trả về object DataBlock. Cùng tìm hiểu xem từng tham số dùng để làm gì nhé
block: Định nghĩa xem Dataloader sẽ trả về gì. Do bài toán của chúng ta là bài toán phân lớp nên Dataloader sẽ trả 2 thứ: ảnh và label tương ứng của nó.get_y: lấy label từ tên file như thế nào. Label của mỗi bức ảnh là 1 phần trong tên file của nó. fastai cung cấp class RegexLabeller sử dụng regular expression để tách label từ tên file. VD:
RegexLabeller(r'/(.+)_\d+.tif$')('dibas_images/Lactobacillus.delbrueckii_0019.tif')
![]()
splitter: chia dataset thành 2 tập train/validationitem_tfms: do ảnh ở các kích thước khác nhau nên cần resize lại thành cùng kích thước mới có thể đóng gói thành từng batch.
Sau khi có Datablock thì chỉ cần chạy:
dls = dblock.dataloaders(source=fns, bs=16)
cùng với các tham số: nguồn dữ liệu (list các file ảnh) và batch size. Phương thức trên sẽ trả về object Dataloaders. Như tên gọi của nó, Dataloaders bao gồm nhiều Dataloader (1 train và 1 validation). Mọi người có thể index vào dls để truy cập các Dataloader: dls[0], dls[1].
Ta có thể kiểm tra xem bộ dữ liệu có bao nhiêu class:
print(dls.vocab)
Training
Việc luyện mô hình được xử lý bằng class Learner. Với bài toán phân lớp các bạn có thể tạo Learner bằng hàm cnn_learner:
learn = cnn_learner(dls, resnet50, metrics=accuracy)
Các tham số bao gồm:
- Dataloaders
- KIến trúc CNN. Ở đây mình dùng Resnet50 nhưng mọi người có thể dùng các mạng CNN pretrain có sẵn trên torchvision
- List các metrics
Khi sử dùng mô hình pretrain, learner sẽ tự thêm các 1 số lớp Linear ở cuối phần CNN
learn.model
(1): Sequential(
(0): AdaptiveConcatPool2d(
(ap): AdaptiveAvgPool2d(output_size=1)
(mp): AdaptiveMaxPool2d(output_size=1)
)
(1): Flatten(full=False)
(2): BatchNorm1d(4096, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(3): Dropout(p=0.25, inplace=False)
(4): Linear(in_features=4096, out_features=512, bias=False)
(5): ReLU(inplace=True)
(6): BatchNorm1d(512, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(7): Dropout(p=0.5, inplace=False)
(8): Linear(in_features=512, out_features=33, bias=False)
)
Mặc định phần trọng số của CNN sẽ đóng băng và không update trong quá trình train.
Việc train model rất đơn giản:
learn.fit_one_cycle(8, 1e-3)
fit_one_cycle(8, 1e-3) sẽ train model trong 8 epoch sử dụng 1-cycle policy. Nếu không muốn sử dụng learning rate scheduler, các bạn có thể dùng phương thức fit
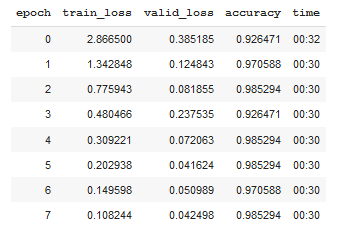
Chỉ sau 8 epoch, accuracy đã đạt 98.5%. Giờ ta sẽ phá băng để train phần CNN:
learn.unfreeze()
learn.fit_one_cycle(5, 1e-7)
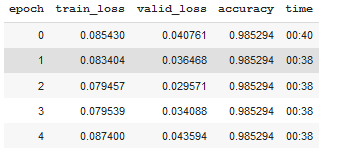
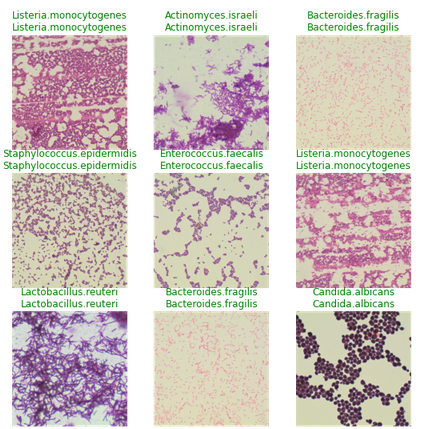
Để xem kết quả của mô hình, mọi người có thể chạy learn.show_results()
Toàn bộ quá trình từ load dữ liệu đến train model chỉ mất chưa đến 10 dòng code
dblock = DataBlock(blocks=(ImageBlock, CategoryBlock),
get_y=RegexLabeller(r'/(.+)_\d+.tif$'),
splitter=RandomSplitter(valid_pct=0.1),
item_tfms=[Resize(512)])
dls = dblock.dataloaders(source=fns, bs=16)
learn = cnn_learner(dls, resnet50, metrics=accuracy)
learn.fit_one_cycle(8, 1e-3)
learn.unfreeze()
learn.fit_one_cycle(5, 1e-7)
Lời kết
Trên đây, mình đã hướng dẫn các bạn cài đặt cài đặt mô hình phân loại vi khuẩn với độ chính xác 98.5% bằng thư viện fastai. Chỉ với chưa tới 10 dòng code, ta đã vượt qua kết quả SOTA 97% trên bộ dữ liệu này (các bạn có thể kiểm tra tại đây). Nếu mọi người thấy bài viết có ích, xin hãy để lại cho mình 1 upvode nhé. Cảm ơn mọi người đã quan tâm và hẹn gặp lại trong những bài tiếp theo.
All rights reserved
