[Kaggle Competition -Plant Pathology 2020 - FGVC7] Phân loại bệnh lá cây
Bài đăng này đã không được cập nhật trong 5 năm
Bài viết này mình sẽ viết về cách phân loại bệnh của lá cây dựa dữ liệu mình lấy từ Plant Pathology Challenge của kaggle.
Dataset
Ở đây BTC cung cấp một tập dữ liệu lá được chụp từ cây táo và yêu cầu phải xác định được bệnh của bức ảnh đó. Tập ảnh bao gồm 4 loại lá : healthy, scab, rust và có nhiều hơn một loại bệnh. Dataset bao gồm:
-
file train.csv
Trong file train.csv chứa 5 cột: image_id, combinations, healthy, rust, scab
-
images
Folder images chứa ảnh tập train và test dưới định dạng jpg
-
test.csv
Trong file test.csv chứa: image_id
Preprocessing
import thư viện đã nào
import os
import gc
import re
import cv2
import math
import numpy as np
import scipy as sp
import pandas as pd
import tensorflow as tf
from keras.utils import plot_model
import tensorflow.keras.layers as L
from keras.utils import model_to_dot
import tensorflow.keras.backend as K
from tensorflow.keras.models import Model
import seaborn as sns
from tqdm import tqdm
import matplotlib.cm as cm
from sklearn import metrics
import matplotlib.pyplot as plt
from sklearn.utils import shuffle
from sklearn.model_selection import train_test_split
tqdm.pandas()
import plotly.express as px
import plotly.graph_objects as go
import plotly.figure_factory as ff
from plotly.subplots import make_subplots
np.random.seed(0)
tf.random.set_seed(0)
import warnings
warnings.filterwarnings("ignore")
Nhiều quá nhưng mà vẫn phải import thôi.
IMAGE_PATH = "../input/plant-pathology-2020-fgvc7/images/"
TEST_PATH = "../input/plant-pathology-2020-fgvc7/test.csv"
TRAIN_PATH = "../input/plant-pathology-2020-fgvc7/train.csv"
SUB_PATH = "../input/plant-pathology-2020-fgvc7/sample_submission.csv"
sub = pd.read_csv(SUB_PATH)
test_data = pd.read_csv(TEST_PATH)
train_data = pd.read_csv(TRAIN_PATH)
LABEL_COLS = ['healthy', 'multiple_diseases', 'rust', 'scab']
Đầu tiên chúng ta sẽ kiểm tra xem trong tập train sẽ bao gồm những gì đã nhé:
train_data.head()
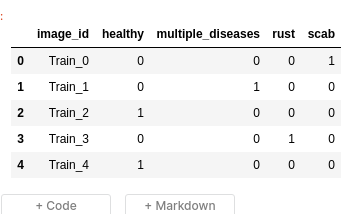
Hình 1: tập train
Như ở hình một tập train của chúng ta bao gồm 4 labels là các tình trạng của lá táo mà chúng ta phải nhận dạng được khi đưa vào được một bức ảnh bất kỳ: 'healthy', 'multiple_diseases', 'rust', 'scab'. Sau đây chúng ta thử kiểm tra xem tỉ lệ mắc bệnh và healthy của tập training như thế nào nhé.
_, axes = plt.subplots(ncols=4, nrows=1, constrained_layout=True, figsize=(10, 3))
for ax, column in zip(axes, LABEL_COLS):
train_data[column].value_counts().plot.bar(title=column, ax=ax)
plt.show()
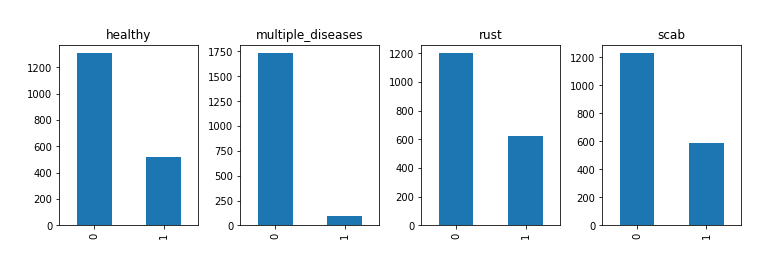
Hình 2: Tỷ lệ các class
plt.title('Label dist')
train_data[LABEL_COLS].idxmax(axis=1).value_counts().plot.bar()
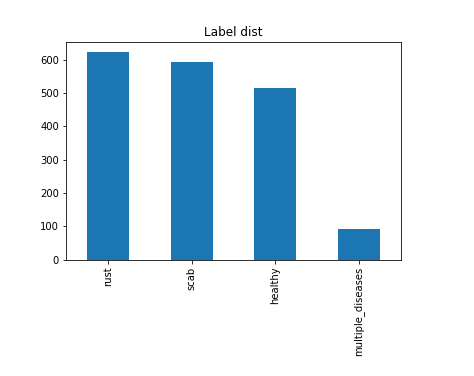
Hình 3: Label distribution
Dựa vào 2 hình trên chúng ta có thể thấy số lá cây bị nhiễm nhiều loại bệnh cùng một lúc cực ít còn tỉ lệ 3 loại còn lại thi khá là đồng đều nhau. Để loại bỏ imblance data thì chúng ta sẽ tăng số lượng ảnh cho "multiple diseases" bằng cách sử dụng các phương pháp Augment data.
Visualize Images
Tiếp theo là load ảnh để check xem hình thật của chúng ta thôi nào.
def load_image(image_id):
file_path = image_id + ".jpg"
image = cv2.imread(IMAGE_PATH + file_path)
return cv2.cvtColor(image, cv2.COLOR_BGR2RGB)
train_images = train_data["image_id"].progress_apply(load_image)
Show thử ảnh đầu xem sao.
plt.imshow(train_images[0])
 Hình 3: test image
Hình 3: test image
Show hình ảnh từng loại label trong 4 labels: 'healthy', 'multiple_diseases', 'rust', 'scab'.
def visualize_leaves(cond_cols=["healthy"]):
cond_list = []
for col in cond_cols:
if col == "healthy":
cond_list.append("healthy==1")
if col == "scab":
cond_list.append("scab==1")
if col == "rust":
cond_list.append("rust==1")
if col == "multiple_diseases":
cond_list.append("multiple_diseases==1")
data = train_data.loc[:100]
for cond in cond_list:
data = data.query(cond)
images = train_images.loc[list(data.index)]
cols, rows = 3, min([3, len(images)//3])
fig, ax = plt.subplots(nrows=rows, ncols=cols, figsize=(30, rows*20/3))
for col in range(cols):
for row in range(rows):
ax[row, col].imshow(images.loc[images.index[row*2+col]])
plt.show()
Những loại lá health sẽ có hình dáng như nào ta?
visualize_leaves(cond_cols=["healthy"])
 Hình 4: Healthy
Hình 4: Healthy
visualize_leaves(cond_cols=["scab"])

Hình 5: scab
pre-processing
Detect Canny và crop images
def crop_image_with_canny(img):
emb_img = img.copy()
height, width, channels = img.shape
edges = cv2.Canny(img, 100, 255)
edge_coors = []
for i in range(edges.shape[0]):
for j in range(edges.shape[1]):
if edges[i][j] != 0:
edge_coors.append((i, j))
row_min = edge_coors[np.argsort([coor[0] for coor in edge_coors])[0]][0]
row_max = edge_coors[np.argsort([coor[0] for coor in edge_coors])[-1]][0]
col_min = edge_coors[np.argsort([coor[1] for coor in edge_coors])[0]][1]
col_max = edge_coors[np.argsort([coor[1] for coor in edge_coors])[-1]][1]
new_img = img[row_min:row_max, col_min:col_max]
return new_img
Do bức ảnh có kích thước khá lớn nên mình sẽ cropped ảnh bớt đi. Ở đây mình sử dụng Canny để detect cạnh sau đó crop ảnh theo. Test thử 1 ảnh được crop xem như nào nha?
plt.imshow(train_images[80])

Hình 6: Ảnh trước khi cropped
img = crop_image_with_canny(train_images[80])
plt.imshow(img)

Hình 7: ảnh sau khi cropped
Ở 2 hình trên chúng ta có thể thấy ảnh đã được cắt bớt đi nhưng phần lá cây vẫn còn nguyên vẹn  ).
).
Augment dữ liệu.
import albumentations
from albumentations import RandomCrop, Compose, HorizontalFlip, VerticalFlip, OneOf
from albumentations.core.transforms_interface import DualTransform
from albumentations.augmentations import functional as F
def augment(aug, image):
'''
image augmentation
aug : augmentation from albumentations
'''
aug_img = aug(image=image)['image']
return aug_img
def VH_augment(image):
'''
Vertical and horizontal flip image
'''
image = HorizontalFlip(p=1)(image=image)['image']
image = VerticalFlip(p=1)(image=image)['image']
return image
def strong_aug(p=1.0):
'''
4D - augmentations
'''
return OneOf([
HorizontalFlip(p=0.33),
VerticalFlip(p=0.33),
Compose([HorizontalFlip(p=1),
VerticalFlip(p=1)], p=0.33)
], p=1)
plt.imshow(train_images[10])
 Hình 8: ảnh trước khi augment
Hình 8: ảnh trước khi augment
aug = strong_aug(p=1.0)
image = augment(aug, train_images[10])
 Hình 9: ảnh sau khi augment
Hình 9: ảnh sau khi augment
Data Generator
Mọi người có thể đọc bài viết này của mình để tham khảo các custom data generator nhé.
class DataGenerator(Sequence):
def __init__(self,
img_paths,
labels,
batch_size=32,
dim=(224, 224),
n_channels=3,
n_classes=4,
add_crop=False,
augmentation=True,
shuffle=False):
self.dim = dim
self.batch_size = batch_size
self.labels = labels
self.img_paths = img_paths
self.n_channels = n_channels
self.n_classes = n_classes
self.shuffle = shuffle
self.augmentation = augmentation
self.add_crop = add_crop
self.img_indexes = np.arange(len(self.img_paths))
self.on_epoch_end()
def __len__(self):
'Denotes the number of batches per epoch'
return int(np.floor(len(self.img_paths) / self.batch_size))
def __getitem__(self, index):
'Generate one batch of data'
# Generate indexes of the batch
indexes = self.indexes[index*self.batch_size:(index+1)*self.batch_size]
# Find list of IDs
list_IDs_temps = [self.img_paths[k] for k in indexes]
# Generate data
X, y = self.__data_generation(list_IDs_temps)
return X, y
def on_epoch_end(self):
'Updates indexes after each epoch'
self.indexes = np.arange(len(self.img_paths))
if self.shuffle == True:
np.random.shuffle(self.indexes)
def __data_generation(self, img_indexes):
X = np.empty((self.batch_size, *self.dim, self.n_channels))
y = []
for i, ID in enumerate(img_indexes):
img = cv2.imread(ID)
img = cv2.cvtColor(img, cv2.COLOR_BGR2RGB)
if self.add_crop == True:
img = crop_image_with_canny(img)
img = cv2.resize(img, self.dim)
if self.augmentation == True:
aug = strong_aug(p=1.0)
image = augment(aug, img)
X[i, ] = img/255
y.append(self.labels[i])
y = np.argmax(y, axis=1)
return X, keras.utils.to_categorical(y, num_classes=4)
Tạo train_gen và val_gen trước khi đưa vào training.
def format_path(st):
return "../input/plant-pathology-2020-fgvc7/images/" + st + '.jpg'
test_paths = test_data.image_id.apply(format_path).values
train_paths = train_data.image_id.apply(format_path).values
train_labels = np.float32(train_data.loc[:, 'healthy':'scab'].values)
train_paths, valid_paths, train_labels, valid_labels =\
train_test_split(train_paths, train_labels, test_size=0.15, random_state=2020)
IMG_SIZE = 512
BATCH_SIZE = 8
N_CHANNELS = 3
train_gen = DataGenerator(
train_paths, train_labels, dim=(IMG_SIZE,IMG_SIZE), batch_size=BATCH_SIZE, shuffle=False)
val_gen = DataGenerator(
valid_paths, valid_labels, dim=(IMG_SIZE,IMG_SIZE), batch_size=BATCH_SIZE, shuffle=False)
Training
Define model
model = Sequential()
model.add(Conv2D(32, kernel_size=(5,5),activation='relu', input_shape=(img_size, img_size, 3)))#, kernel_regularizer=l2(reg)))
model.add(BatchNormalization(axis=-1,center=True,scale=False))
model.add(Conv2D(64, kernel_size=(5,5),activation='relu'))#,kernel_regularizer=l2(reg)))
model.add(BatchNormalization(axis=-1,center=True,scale=False))
model.add(MaxPooling2D(pool_size=(2,2), padding='SAME'))
model.add(Dropout(.25))
model.add(Conv2D(32, kernel_size=(3,3),activation='relu'))#,kernel_regularizer=l2(reg)))
model.add(BatchNormalization(axis=-1,center=True,scale=False))
model.add(Conv2D(64, kernel_size=(3,3),activation='relu'))#,kernel_regularizer=l2(reg)))
model.add(BatchNormalization(axis=-1,center=True,scale=False))
model.add(MaxPooling2D(pool_size=(2,2), padding='SAME'))
model.add(Dropout(.25))
model.add(Conv2D(64, kernel_size=(5,5),activation='relu'))#, kernel_regularizer=l2(reg)))
model.add(BatchNormalization(axis=-1,center=True,scale=False))
model.add(Conv2D(128, kernel_size=(5,5),activation='relu',))#kernel_regularizer=l2(reg)))
model.add(BatchNormalization(axis=-1,center=True,scale=False))
model.add(MaxPooling2D(pool_size=(2,2), padding='SAME'))
model.add(Dropout(.25))
model.add(Conv2D(64, kernel_size=(3,3),activation='relu'))#,kernel_regularizer=l2(reg)))
model.add(BatchNormalization(axis=-1,center=True,scale=False))
model.add(Conv2D(128, kernel_size=(3,3),activation='relu'))#,kernel_regularizer=l2(reg)))
model.add(BatchNormalization(axis=-1,center=True,scale=False))
model.add(MaxPooling2D(pool_size=(2,2), padding='SAME'))
model.add(Dropout(.25))
model.add(Flatten())
model.add(Dense(300,activation='relu'))
model.add(BatchNormalization(axis=-1,center=True,scale=False))
model.add(Dropout(.25))
model.add(Dense(200,activation='relu'))
model.add(BatchNormalization(axis=-1,center=True,scale=False))
model.add(Dropout(.25))
model.add(Dense(100,activation='relu'))
model.add(BatchNormalization(axis=-1,center=True,scale=False))
model.add(Dropout(.25))
model.add(Dense(4,activation='softmax'))
Compile model
model.summary()
from keras.preprocessing.image import ImageDataGenerator
model.compile(optimizer='adam',
loss='categorical_crossentropy',
metrics=['accuracy']
)
reduce_learning_rate = ReduceLROnPlateau(monitor='loss',
factor=0.1,
patience=2,
cooldown=2,
min_lr=0.000001,
verbose=1)
callbacks = [reduce_learning_rate]
training
model.fit_generator(train_gen, epochs=50,validation_data=val_gen
,verbose=1,callbacks=callbacks,use_multiprocessing=False,
shuffle=True)
predict và tạo submission
test_img=[]
path='/kaggle/input/plant-pathology-2020-fgvc7/images'
for im in tqdm(test['image_id']):
im=im+".jpg"
final_path=os.path.join(path,im)
img=cv2.imread(final_path)
img=cv2.resize(img,(224, 224))
img=img.astype(('float32'))
test_img.append(img)
y_pred=model.predict(test_img)
print(y_pred)
 Hình: y_pred
Hình: y_pred
submission.loc[:,'healthy':'scab']=y_pred
submission.to_csv('submission.csv',index=False)
Sau khi hoàn tất thì tải file submission.csv về nộp thôi 
Kết Luận
Bài viết này mục đích là để hướng dẫn các bước khi join kaggle competion và custom DataGenerator do vậy kết quả predict không chính xác lắm. Mọi người có thể tự custom lại model và preprocessing data theo ý mình để đạt được kết quả tốt hơn nhé. Cảm ơn mọi người đã đọc bài của mình. Rất mong nhận được ý kiến đóng góp của mọi người.
Reference
All rights reserved
