Data visualization trong Machine Learning - Phần 2
Bài đăng này đã không được cập nhật trong 7 năm
Chào mọi người, trong phần đầu của series Data visualization trong Machine Learning mình đã giới thiệu một vài vấn đề cơ bản như thế nào là Data visualization, ứng dụng của Data visualization trong Machine Learning, một số dạng biểu đồ thường gặp. Trong phần tiếp theo này mình sẽ giới thiệu một số biểu đồ cụ thể thường được sử dụng trong giai đoạn data preprocessing (tiền xử lý dữ liệu) trong bài toán Machine Learning.
Note 1: Một số thư viện cần sử dụng bao gồm:
- Pandas & Numpy: Thư viện để phân tích, xử lý dữ liệu thô.
- Scikit-learn: Thư viện về machine learning cơ bản nhất của Python.
- Matplotlib, seaborn, yellowbrick: Các thư viện phục vụ việc mô hình hóa dữ liệu, trong đó seaborn và yellowbrick được phát triển base trên matplotlib nhưng dễ sử dụng hơn rất nhiều. Đặc biệt trong phần đầu của series này mình sẽ chủ yếu sử dụng yellowbrick vì đây là thư viện đặc biệt dễ dùng và hiệu quả rất tốt khi kết hợp cùng scikit-learn.
- Mọi người có thể dễ dàng cài đặt các thư viện trên thông qua pip với cú pháp: pip install tên_thư_viện.
Note 2: Trong series này mình sẽ sử dụng bộ dữ liệu về ung thư vú mọi người có thể download tại đây. Bộ dữ liệu này bao gồm hai phân lớp được gán nhãn "2" là những bệnh nhân dấu hiệu nhẹ và "4" là những bệnh nhân ác tính. Dữ liệu được load và xử lý cơ bản như sau:
# Import lib import pandas as pd from sklearn import preprocessing df = pd.read_csv('../input/breast-cancer-wisconsin.data.csv') # Load breast cancer data from csv file df = df.replace('?', np.NaN) # Replace '?' by null value classes = df.Class.value_counts().keys() # Get classes of data set le = preprocessing.LabelEncoder() df['Class'] = le.fit_transform(df['Class']) #Label Encoder classes # List features for visualization features = ['Clump Thickness', 'Uniformity of Cell Size', 'Uniformity of Cell Shape', 'Marginal Adhesion', 'Single Epithelial Cell Size', 'Bare Nuclei', 'Bland Chromatin', 'Normal Nucleoli', 'Mitoses'] X = df[features].fillna(0) # Data values
Note 3: Với mỗi dạng biểu đồ mình sẽ trình bày các nội dung:
- Ý nghĩa: Nội dung mà biểu đồ đó thể hiện, từ biểu đồ ta có thể nhận xét điều gì về tập data đang xét.
- Tham số: Các tham số cơ bản cần cài đặt của biểu đồ.
- Lưu ý (nếu có): Có thể là các lưu ý hoặc cơ bản về thuật toán để xử lý data, thuật toán để vẽ biểu đồ.
- Code: Source code tham khảo để vẽ biểu đồ.
Biểu diễn các đặc trưng của dữ liệu:
1. Biểu diễn dữ liệu khuyết thiếu:
Dạng 1:
Ý nghĩa: Các giá trị khuyết thiếu sẽ được biểu diễn bằng màu vàng, từ biểu đồ ta có thể quan sát được mức độ khuyết thiếu giá trị của mỗi feature.
Tham số:
- data (type: dataframe): df.isnull() nếu giá trị tại ô đang xét là null thì sẽ return True, ngược lại return False.
- yticklabels (type: boolean): Nếu set là True trục y sẽ biểu diễn miền giá trị của từng feature, nếu là False trục y sẽ được ẩn.
- cbar (type: boolean): Nếu set là True color bar của biểu đồ sẽ được vẽ, nếu là False color bar sẽ được ẩn.
- cmap (type: color map hoặc list color): Bảng màu của đồ thị.
Code:
import seaborn as sns
%matplotlib inline
sns.heatmap(data=df.isnull(), yticklabels=False, cbar =False, cmap = 'viridis')
Dạng 2:
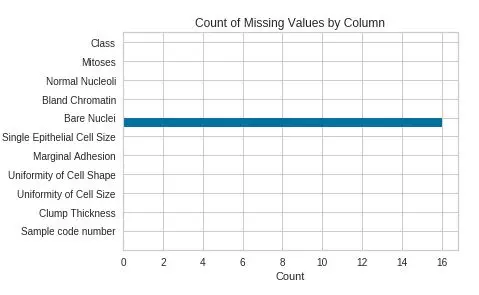
Ý nghĩa: Biểu diễn số lượng giá trị khuyết thiếu của mỗi feature, như ví dụ trên có thể thấy cột "Bare Nuclei" có 16 ô bị khuyết thiếu giá trị.
Tham số:
- feature (type: array): Mảng các feture cần biểu diễn.
- df (type: dataframe): Dataframe cần biểu diễn.
Code:
from yellowbrick.contrib.missing import MissingValuesBar
viz = MissingValuesBar(features=df.columns)
viz.fit(df)
viz.poof()
2. Biểu diễn sự cân bằng của các phân lớp
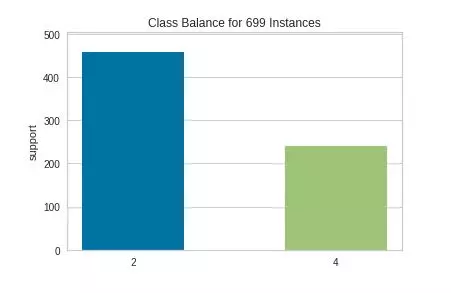
Ý nghĩa: Từ biểu đồ ta có thể thấy được số lượng mẫu của mỗi phân lớp, sự cân bằng/mất cân bằng giữa các phân lớp trong bộ dữ liệu. Như ví dụ trên có thể thấy số lượng mẫu bệnh nhân ác tính là khá thấp so với số lượng mẫu bệnh nhân dấu hiệu nhẹ.
Tham số:
- feature (type: list): Các phân lớp được biểu diễn ở trục x, nếu tham số này không được chỉ định thì function sẽ mặc định lấy cột labels trong bộ dữ liệu.
- y_train (type: array): một array hoặc list các giá trị của phân lớp có số chiều là (n,).
Lưu ý:
- Với ClassBalance giá trị của các phân lớp phải là các giá trị rời rạc nên ta bắt buộc phải thực hiện bước Label Encoder cho cột Label.
Code:
from yellowbrick.target import ClassBalance
visualizer = ClassBalance(labels=classes)
visualizer.fit(y_train=df['Class'])
visualizer.poof()
3. Biểu diễn miền giá trị, giá trị ngoại lai của các features
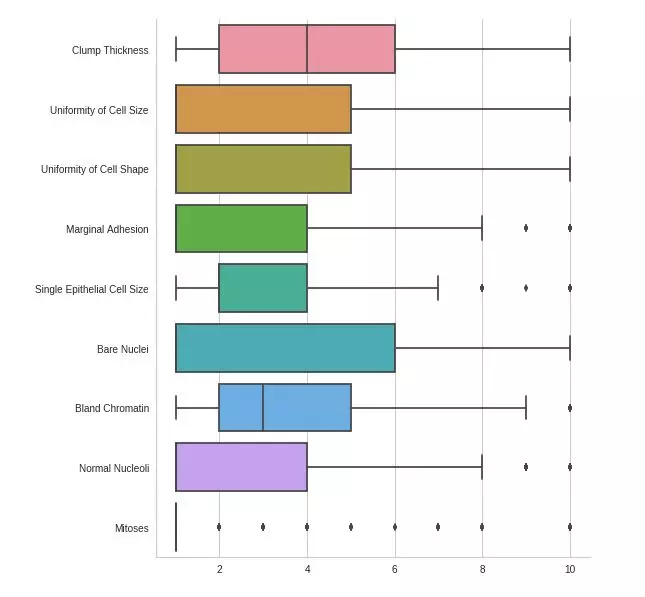
Ý nghĩa: Từ biểu đồ ta có thể thấy được giá trị min, giá trị max, miền giá trị, giá trị ngoại lai của các feature trong bộ dữ liệu.
Tham số:
- data (type: data frame): Tập dữ liệu đầu vào.
- orient ("v" hoặc "h"): Chiều của biểu đồ, "h" biểu đồ sẽ được vẽ theo chiều ngang, "v" biểu đồ sẽ được vẽ theo chiều dọc.
- kind (“point”, “bar”, “strip”, “swarm”, “box”, “violin”, or “boxen”): Dạng của biểu đồ sẽ được vẽ (mọi người có thể thay đổi tham số này để có được các dạng đồ thị khác khá là hay ho
 ).
).
Code:
import seaborn as sns
sns.catplot(data=df[features], orient="h", kind="box")
4. Biểu đồ Parallel Coordinates
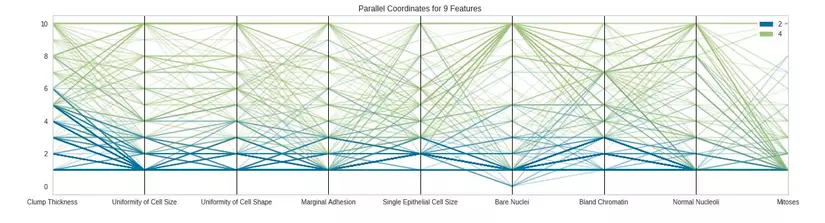
Ý nghĩa: Đây là một dạng biểu đồ đa biến, dạng biểu đồ này sẽ cho phép chúng ta so sánh nhiều features với nhau và mối quan hệ giữa chúng. Ngoài ra nó còn thể hiện sự chồng chéo hoặc phân tách giữa các phân lớp. Như ví dụ trên đây ta có thể thấy rằng đa số các features nếu có giá trị cao vượt quá 6 thì sẽ rơi vào phân lớp 4 (bệnh nhân ác tính).
Tham số:
- feature (type: list/array): Mảng các features cần biểu diễn.
- sample (type: int/float): Có bao nhiêu mẫu trong tập dữ liệu sẽ được biểu diễn, nếu là float sẽ tương đương với phần trăm (0.5 = 50%) nếu là int sẽ là số lượng mẫu cụ thể được chỉ định.
- normalize (string hoặc None): kỹ thuật normalization sẽ được sử dụng ( ‘minmax’, ‘maxabs’, ‘standard’, ‘l1’, ‘l2’), default: None
- X (type: dataframe): Tập dữ liệu đầu vào.
- y (type: array): một array hoặc list các giá trị của phân lớp có số chiều là (n,).
Code:
from yellowbrick.features import ParallelCoordinates
visualizer = ParallelCoordinates(classes=classes, features=features, sample=0.5)
visualizer.fit_transform(X=X, y=df['Class'])
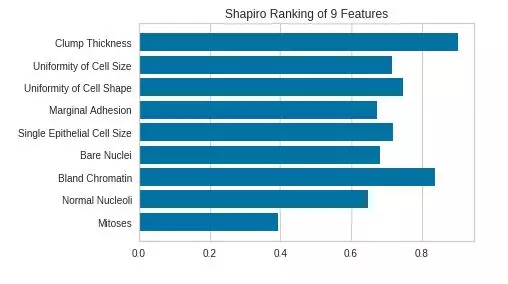
5. Ranking Features
Dạng 1: Rank 1D
Ý nghĩa: Đánh giá đơn lẻ từng features dựa trên các thuật toán ranking từ đó có thể lựa chọn các features để đưa vào training model.
Tham số:
- feature (type: list/array): Mảng các features cần biểu diễn.
- algorithm ('shapiro'): thuật toán ranking, default: shapiro-wilk.
Lưu ý:
- shapiro-wilk: là một kiểm nghiệm thống kê phân phối chuẩn của một biến số. Kết quả của phép kiểm nghiệm này sẽ cho ra một chỉ số p-value, nếu p-value > 0.05 thì khả năng biến có phân phối chuẩn.
- Trong Rank1D toàn bộ dữ liệu đưa vào ranking bắt buộc phải đồng nhất về kiểu. Trong ví dụ dưới đây toàn bộ data trong tập dữ liệu đầu vào sẽ được ép về float
Code:
from yellowbrick.features import Rank1D
# Instantiate the 1D visualizer with the Sharpiro ranking algorithm
visualizer = Rank1D(features=features, algorithm='shapiro')
visualizer.fit_transform(X=X.astype(float), y=df['Class'])
visualizer.poof()
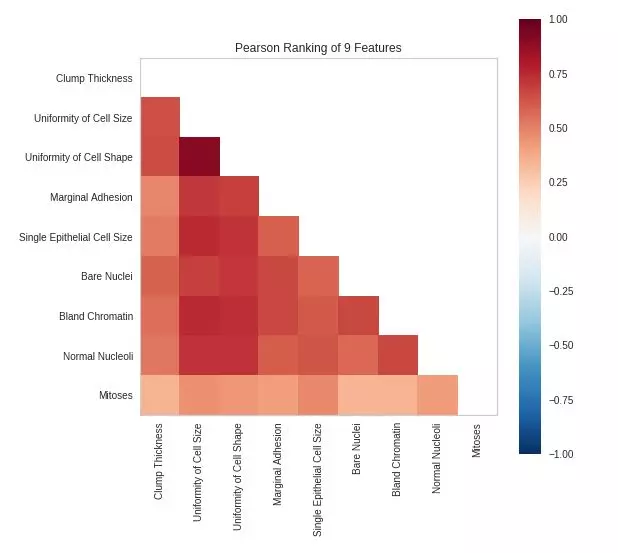
Dạng 2: Rank 2D
Ý nghĩa: Đánh giá từng cặp hai features dựa trên các thuật toán ranking.
Tham số:
- feature (type: list/array): Mảng các features cần biểu diễn.
- algorithm ('pearson', 'covariance', 'spearman'): thuật toán ranking, default: pearson.
Lưu ý:
-
pearson: Là một chỉ số thống kê đo lường mức độ tương quan giữa hai biến số (hệ số tương quan), hệ số tương quan nằm trong khoảng [-1, 1], nếu hệ số này = 0 tức là 2 biến số không có liên hệ gì với nhau. Hệ số tương quan pearson chỉ hợp lý nếu hai biến số đang xét đều tuân theo luật phân phối chuẩn.
-
spearman: Nếu x và y không tuân theo luật phân phối chuẩn, chúng ta phải sử dụng một hệ số tương quan khác tên là Spearman, một phương pháp phân tích phi tham số. Hệ số này được ước tính bằng cách biến đổi hai biến số x và y thành thứ bậc (rank), và xem độ tương quan giữa hai dãy số bậc.
-
covariance: Là thước đo độ biến thiên cùng nhau của hai biến số ngẫu nhiên. Nếu hai biến số đang xét cùng tăng hoặc cùng giảm thì covariance tăng. Nếu một biến nằm trên kỳ vọng, một biến nằm dưới kỳ vọng thì covariance sẽ có giá trị âm.
-
Tương tự với Rank1D toàn bộ dữ liệu đầu vào của Rank2D cũng cần ép về một kiểu đồng nhất.
Code:
from yellowbrick.features import Rank2D
# Instantiate the visualizer with the Covariance ranking algorithm
visualizer = Rank2D(features=features, algorithm='pearson')
visualizer.fit_transform(X=X.astype(float), y=df['Class'])
visualizer.poof()
Trên đây mình đã giới thiệu các dạng biểu đồ thường xử dụng trong quá trình tiền xử lý dữ liệu. Trong phần tiếp theo của series mình sẽ giới thiệu với mọi người một số dạng biểu đồ biểu diễn sự phân bố của dữ liệu và một số dạng biểu đồ phục vụ bài toán phân lớp (classification).
All rights reserved